资讯专栏INFORMATION COLUMN

此篇文章关键给大家介绍了应用Python脚本制作获取基因组测序指定位置编码序列的实例详细说明,感兴趣的小伙伴值得借鉴参考一下,也希望能有一定的帮助,祝愿大家多多的发展,尽早涨薪
前言
在基因组分析中,大家常常会有这样一个要求,便是在一个fasta文件中获取某些编码序列出去。有时候这种编码序列注定是完备的编码序列,而有时候只是为原fasta文件中某一段编码序列中的一部分。尤其是当信息量许多时,应用人眼来挑选编码序列会很费劲,因此这个时候大家就可以用编程逻辑去完成了。
比如这里在百度云盘配件中给出了某种群全基因组序列(0-refer/Bacillus_subtilis.str168.fasta),及其基因组测序gff注解文件(0-refer/Bacillus_subtilis.str168.gff)。
假定在这儿对于该种群进行分析,根据gff注解文档中的基因功能叙述字段名,再加上对相关信息的查阅等,精准定位到了一部分特定的基因。
下面我们期待基于gff文件中对这种遗传基因部位的描写,在这一基因组序列fasta文件里将这种遗传基因寻找并分离出来,获得了一个新的fasta文件,新文件上只包括目地基因序列。
请使用python3撰写1个能够实现这个功能的脚本制作。
示例
一个示例脚本如下(可参见网盘附件“seq_select1.py”)。
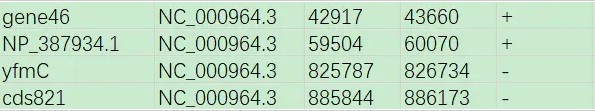
为了实现以上目的,我们首先需要准备一个txt文件(以下称其为list文件,示例list.txt可参见网盘附件),基于gff文件中所记录的基因位置信息,填入类似以下的内容(列与列之间以tab分隔)。
#下列内容保存到list.txt
gene46 NC_000964.3 42917 43660+ NP_387934.1 NC_000964.3 59504 60070+ yfmC NC_000964.3 825787 826734- cds821 NC_000964.3 885844 886173-
第1列,给所要获取的新序列命个名称;
第2列,所要获取的序列所在原序列ID;
第3列,所要获取的序列在原序列中的起始位置;
第4列,所要获取的序列在原序列中的终止位置;
第5列,所要获取的序列位于原序列的正链(+)或负链(-)。
之后根据输入文件,即输入fasta文件及记录所要获取序列位置的list文件中的内容,编辑py脚本。
打开fasta文件“Bacillus_subtilis.scaffolds.fasta”,使用循环逐行读取其中的序列id及碱基序列,并将每条序列的所有碱基合并为一个字符串;将序列id及该序列合并后的碱基序列以字典的形式存储(字典样式{'id':'base'})。
打开list文件“list.txt”,读取其中的内容,存储到字典中。字典的键为list文件中的第1列内容;字典的值为list文件中第2-5列的内容,并按tab分割得到一个列表,包含4个字符分别代表list文件中第2-5列的信息)。
最后根据读取的list文件中序列位置信息,在读取的基因组中截取目的基因序列。由于某些基因序列可能位于基因组负链中,需取其反向互补序列,故首先定义一个函数rev(),用于在后续调用得到反向互补序列。在输出序列名称时,还可选是否将该序列的位置信息一并输出(name_detail=True/False)。
<pre class="r"style="overflow-wrap:break-word;font-style:normal;font-variant-ligatures:normal;font-variant-caps:normal;font-weight:400;letter-spacing:normal;orphans:2;text-align:start;text-indent:0px;text-transform:none;widows:2;word-spacing:0px;-webkit-text-stroke-width:0px;text-decoration-style:initial;text-decoration-color:initial;margin-top:0px;margin-bottom:10px;padding:9.5px;border-radius:4px;background-color:rgb(245,245,245);box-sizing:border-box;overflow:auto;font-size:13px;line-height:1.42857;color:rgb(51,51,51);word-break:break-all;border:1px solid rgb(204,204,204);font-family:"Times New Roman";">#!/usr/bin/env python3
#-*-coding:utf-8-*-
#初始传递命令
input_file='Bacillus_subtilis.str168.fasta'
list_file='list.txt'
output_file='gene.fasta'
name_detail=True
##读取文件
#读取基因组序列
seq_file={}
with open(input_file,'r')as input_fasta:
for line in input_fasta:
line=line.strip()
if line[0]=='>':
seq_id=line.split()[0]
seq_file[seq_id]=''
else:
seq_file[seq_id]+=line
input_fasta.close()
#读取列表文件
list_dict={}
with open(list_file,'r')as list_table:
for line in list_table:
if line.strip():
line=line.strip().split('t')
list_dict[line[0]]=[line[1],int(line[2])-1,int(line[3]),line[4]]
list_table.close()
##截取序列并输出
#定义函数,用于截取反向互补
def rev(seq):
base_trans={'A':'T','C':'G','T':'A','G':'C','N':'N','a':'t','c':'g','t':'a','g':'c','n':'n'}
rev_seq=list(reversed(seq))
rev_seq_list=[base_trans[k]for k in rev_seq]
rev_seq=''.join(rev_seq_list)
return(rev_seq)
#截取序列并输出
output_fasta=open(output_file,'w')
for key,value in list_dict.items():
if name_detail:
print('>'+key,'['+value[0],value[1]+1,value[2],value[3]+']',file=output_fasta)
else:
print('>'+key,file=output_fasta)
seq=seq_file['>'+value[0]][value[1]:value[2]]
if value[3]=='+':
print(seq,file=output_fasta)
elif value[3]=='-':
seq=rev(seq)
print(seq,file=output_fasta)
output_fasta.close()</pre>编辑该脚本后运行,输出新的fasta文件“gene.fasta”,其中的序列即为我们所想要得到的目的基因序列。
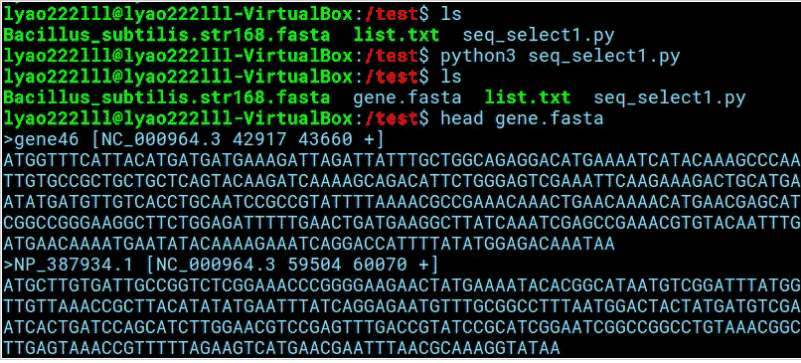
扩展:
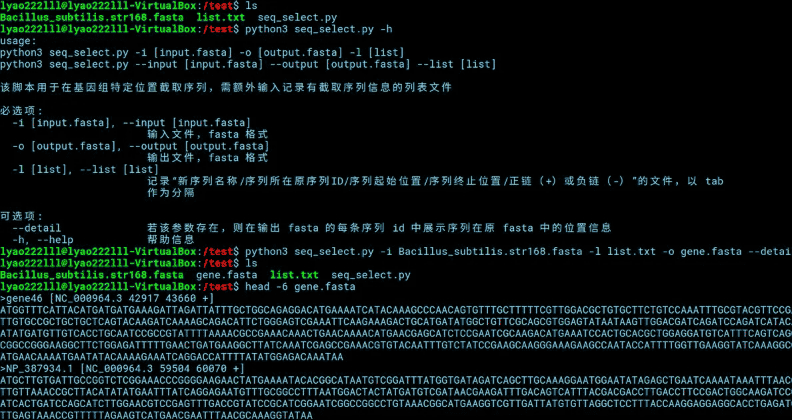
网盘附件“seq_select.py”为添加了命令传递行的python3脚本,可在shell中直接进行目标文件的I/O处理。该脚本可指定输入fasta序列文件以及记录有所需提取序列位置的列表文件,输出的新fasta文件即为提取出的序列。
#!/usr/bin/env python3
#-*-coding:utf-8-*-
#导入模块,初始传递命令、变量等
import argparse
parser=argparse.ArgumentParser(description='n该脚本用于在基因组特定位置截取序列,需额外输入记录有截取序列信息的列表文件',add_help=False,usage='npython3 seq_select.py-i[input.fasta]-o[output.fasta]-l<ul>npython3 seq_select.py--input[input.fasta]--output[output.fasta]--list<ul>')
required=parser.add_argument_group('必选项')
optional=parser.add_argument_group('可选项')
required.add_argument('-i','--input',metavar='[input.fasta]',help='输入文件,fasta格式',required=True)
required.add_argument('-o','--output',metavar='[output.fasta]',help='输出文件,fasta格式',required=True)
required.add_argument('-l','--list',metavar='<ul>',help='记录“新序列名称/序列所在原序列ID/序列起始位置/序列终止位置/正链(+)或负链(-)”的文件,以tab作为分隔',required=True)
optional.add_argument('--detail',action='store_true',help='若该参数存在,则在输出fasta的每条序列id中展示序列在原fasta中的位置信息',required=False)
optional.add_argument('-h','--help',action='help',help='帮助信息')
args=parser.parse_args()
##读取文件
#读取基因组序列
seq_file={}
with open(args.input,'r')as input_fasta:
for line in input_fasta:
line=line.strip()
if line[0]=='>':
seq_id=line.split()[0]
seq_file[seq_id]=''
else:
seq_file[seq_id]+=line
input_fasta.close()
#读取列表文件
list_dict={}
with open(args.list,'r')as list_file:
for line in list_file:
if line.strip():
line=line.strip().split('t')
list_dict[line[0]]=[line[1],int(line[2])-1,int(line[3]),line[4]]
list_file.close()
##截取序列并输出
#定义函数,用于截取反向互补
def rev(seq):
base_trans={'A':'T','C':'G','T':'A','G':'C','a':'t','c':'g','t':'a','g':'c'}
rev_seq=list(reversed(seq))
rev_seq_list=[base_trans[k]for k in rev_seq]
rev_seq=''.join(rev_seq_list)
return(rev_seq)
#截取序列并输出
output_fasta=open(args.output,'w')
for key,value in list_dict.items():
if args.detail:
print('>'+key,'['+value[0],value[1]+1,value[2],value[3]+']',file=output_fasta)
else:
print('>'+key,file=output_fasta)
seq=seq_file['>'+value[0]][value[1]:value[2]]
if value[3]=='+':
print(seq,file=output_fasta)
elif value[3]=='-':
seq=rev(seq)
print(seq,file=output_fasta)
output_fasta.close()适用上述示例中的测试文件,运行该脚本的方式如下。
#python3 seq_select.py-h python3 seq_select.py-i Bacillus_subtilis.str168.fasta-l list.txt-o gene.fasta--detail 源码提取链接:https://pan.baidu.com/s/1kUhBTmpDonCskwmpNIJPkA?pwd=ih9n
提取码:ih9n
文章版权归作者所有,未经允许请勿转载,若此文章存在违规行为,您可以联系管理员删除。
转载请注明本文地址:https://www.ucloud.cn/yun/128450.html
摘要:第三代基因测序技术革新云计算的应用一位准妈妈,在怀孕周时,需要做唐氏儿的筛查,传统唐筛的方式准确率低,如果结果显示危险性高,那么准妈妈还需要做羊膜穿刺等进一步检查。未来组目前已经拥有两台第三代基因测序仪,而未来这一数字将增长至五台。 第三代基因测序技术革新 云计算的应用一位准妈妈,在怀孕12-24周时,需要做唐氏儿的筛查,传统唐筛的方式准确率低,如果结果显示危险性高,那么准妈妈还需要做...
摘要:今天推出了一个名叫的开源工具,用深度神经网络来从测序数据中快速较精确识别碱基变异位点。今天,团队,联合同属于旗下的生命科学兄弟公司,用了两年多时间,研发出了一个名叫的开源工具,专门用深度神经网络来识别结果中测序数据里这些碱基变异位点。 Google今天推出了一个名叫DeepVariant的开源工具,用深度神经网络来从DNA测序数据中快速较精确识别碱基变异位点。学科研究的革命性进展,特别是基因...
摘要:通过选用云主机,基因企业在基因计算环节可以大幅提升产能而普通大众,也能享受成本降低带来的普惠。而客户选用云主机,避免了维护复杂板卡的大量人力物力的投入,缩减了验证平台的维护成本。 摘要: 概括F3经典使用场景 人工智能深度学习客户,推理应用 最近两年,人工智能在全球掀起了巨大的应用热潮,除了互联网巨头,如Google,Facebook,Alibaba之外,涌现出众多的Start up公...
摘要:华为生科云解决方案,由工作流弹性计算云对象云存储线下数据寄送服务四部分组成,为客户提供端到端的解决方案,助力中国科研数据分析,演绎了生物与计算的完美结合。 随着互联网的普及和技术的发展,大数据和云计算已经渗透在人们的生活的各个方面,在金融,零售,能源,交通等领域已经得到广泛应用。而对于生物信息来说,生物的DNA、基因序列、生物芯片等无时无刻不产生新的数据;比如说,DNA测序每年能够产生大约1...
阅读 1170·2023-01-14 11:38
阅读 1162·2023-01-14 11:04
阅读 991·2023-01-14 10:48
阅读 2543·2023-01-14 10:34
阅读 1247·2023-01-14 10:24
阅读 1130·2023-01-14 10:18
阅读 737·2023-01-14 10:09
阅读 821·2023-01-14 10:02